La Paléogénomique : comprendre le passé pour prédire le futur
Caroline Pont

Exploiter le déluge de données génomiques des dernières décennies
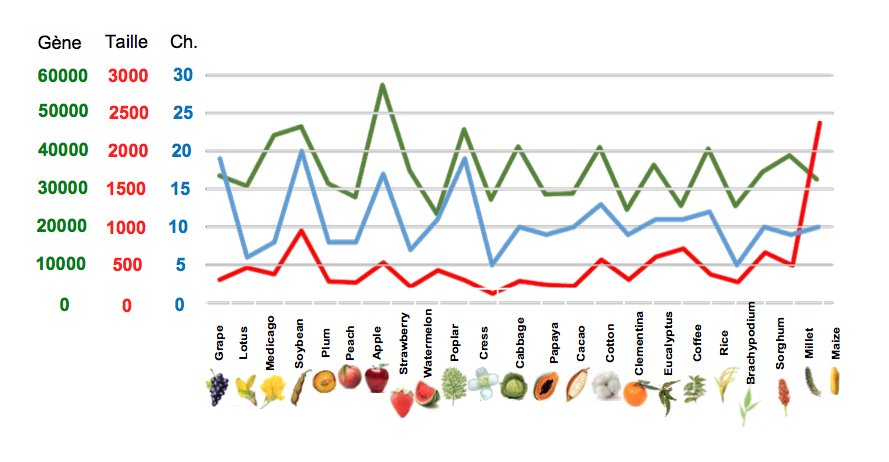
Les plantes constituent la composante fondamentale de notre environnement et génèrent l’essentiel de la biomasse présente à la surface de la terre. Elles représentent aussi la plus grande source de nourriture du règne animal. Leur domestication par l’homme, pour assurer son alimentation, a été déterminante dans sa capacité à peupler la planète. La masse de données génomiques acquises ces 20 dernières années a permis d’améliorer considérablement la compréhension de l’organisation et de la régulation des génomes des plantes à fleurs. Ces dernières, appelées aussi angiospermes, sont constituées de deux grandes familles, les monocotylédones (céréales) et les dicotylédones (légumineuses, crucifères, arbres fruitiers…). A ce jour, plus de 100 génomes de plantes ont été totalement ou partiellement séquencés : le riz, le maïs, le sorgho, le brome, la vigne, l’arabette, le peuplier, la papaye, la luzerne, le soja, le pommier, le concombre…. Ces différents génomes (cf. Figure 1) ont des structures très diverses, de 20 000 à 60 000 gènes, un nombre de chromosomes allant de 5 à plus de 20, des tailles de quelques centaines à plusieurs milliers de mégabases… Toutes ces espèces dérivent d’une espèce ancestrale commune mais quelle est la structure du génome fondateur et permet-il d’élucider les mécanismes qui ont participé à une telle diversité de génomes modernes ? Les recherches menées au sein de l’équipe de recherche PaleoEVO visent à répondre à ces questions.
Reconstruire l’ancêtre des plantes modernes
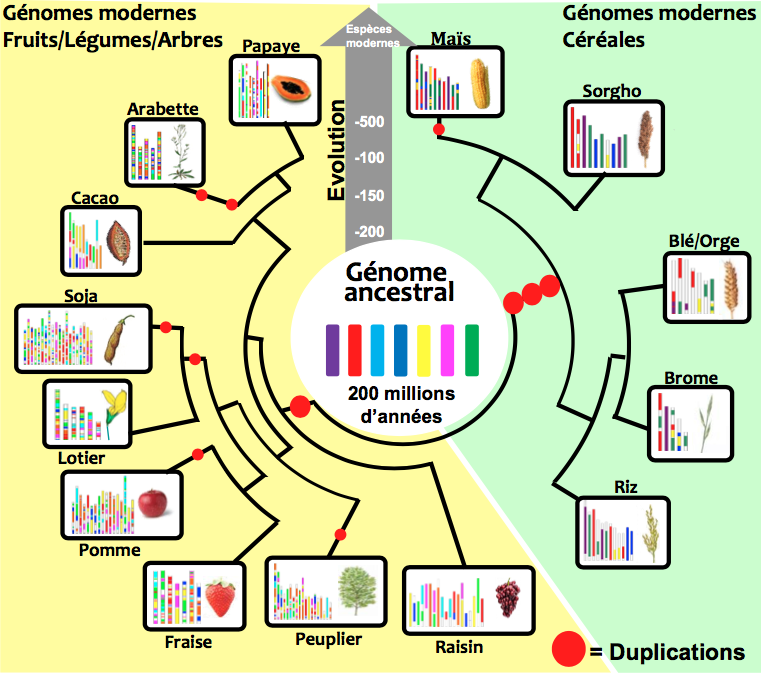
Les chercheurs ont modélisé le génome des ancêtres disparus (on parle de paléogénomique) des plantes à fleurs et ont montré qu’elles dérivent d’ancêtres constitués de 7 chromosomes porteurs de près de 20 000 gènes fondateurs communs à toutes les espèces (Figure 2). Ces gènes fondateurs codent pour des fonctions biologiques de ‘base’, notamment impliquées dans le développement et l’architecture de la plante. Il existe aussi des gènes spécifiques de chaque espèce qui sont engagés dans des fonctions plus ‘spécialisées’, c’est-à-dire la régulation de processus biologiques complexes.
Comprendre l’évolution et la genèse des différentes espèces végétales modernes
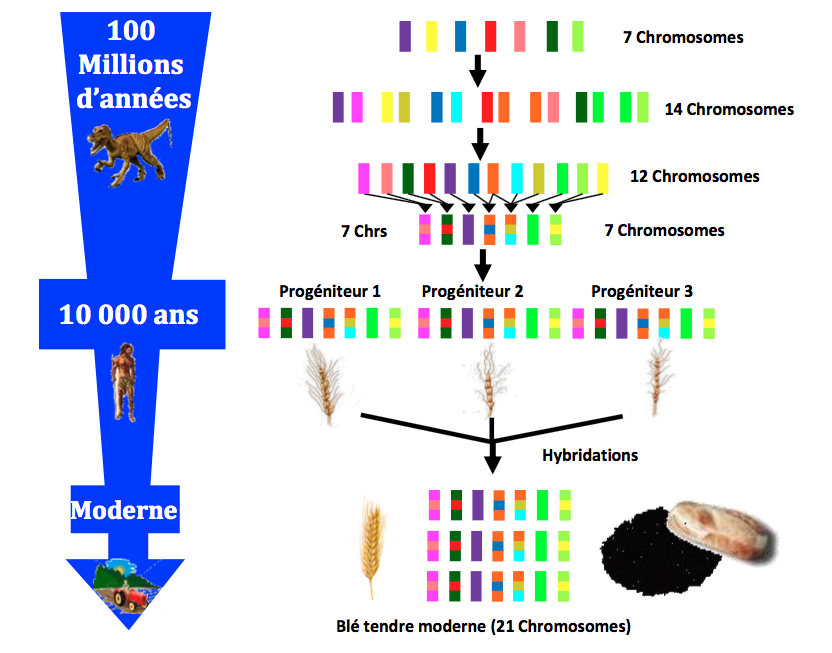
Au cours de l’évolution, les chromosomes ancestraux ont été remaniés et ont fusionné entre eux pour donner naissance à de nouvelles espèces aux génomes distincts. Ainsi, les génomes de plantes modernes apparaissent comme une mosaïque de chromosomes ancestraux réarrangés. Les travaux des chercheurs de l’équipe PaleoEVO ont permis de démontrer que l’apparition de nouvelles espèces de plantes était liée à des fusions de chromosomes et que chaque espèce pouvait être associée à un nombre de fusions chromosomiques distinct (Figure 3).
Elucider les mécanismes de l’adaptation des plantes modernes aux contraintes
Au-delà des réarrangements des chromosomes ancestraux, le doublement du contenu chromosomique (on parle de polyploïdie) a également joué un rôle majeur dans l’évolution et l’adaptation des plantes. En effet, la majorité des plantes, y compris celles cultivées à l’heure actuelle, a connu des évènements de duplication du génome assez récemment, soit quelques millions d’années pour le maïs, le soja, le pommier, le peuplier, la moutarde… D’autres ont conservé des « vestiges » d’événements de duplication plus anciens, plusieurs dizaines de millions d’années pour le riz, le brome, la vigne, la papaye, le sorgho… (Figure 4). Ces duplications ont fourni des copies surnuméraires de gènes qui se sont alors spécialisées. La plante a acquis de nouvelles fonctions biologiques permettant notamment une meilleure adaptation aux contraintes. Cette extrême variabilité et plasticité des gènes issus des duplications constituent toujours à l’heure actuelle, un atout majeur, permettant aux plantes de s’adapter en permanence aux conditions environnementales changeantes.
Faciliter le transfert des connaissances entre espèces végétales modernes
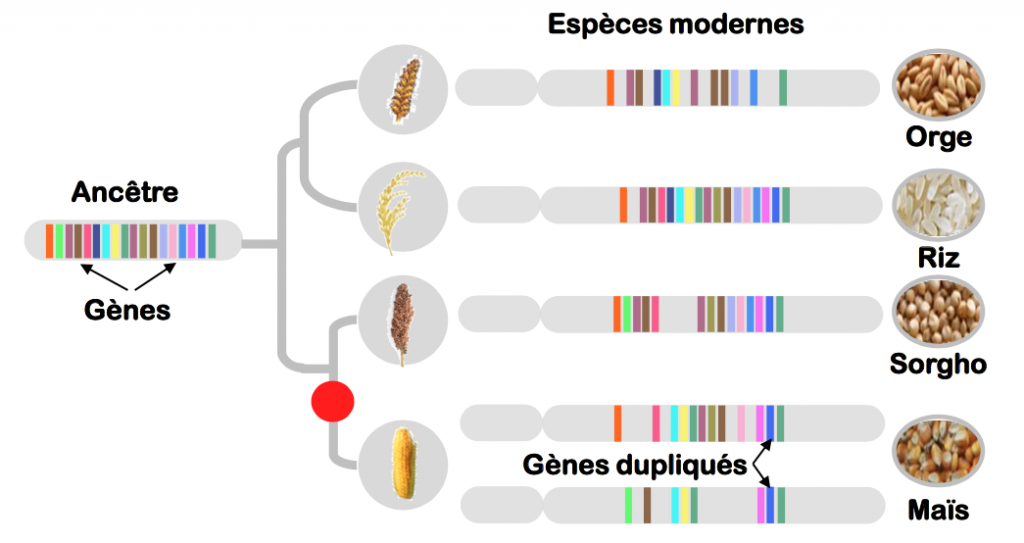
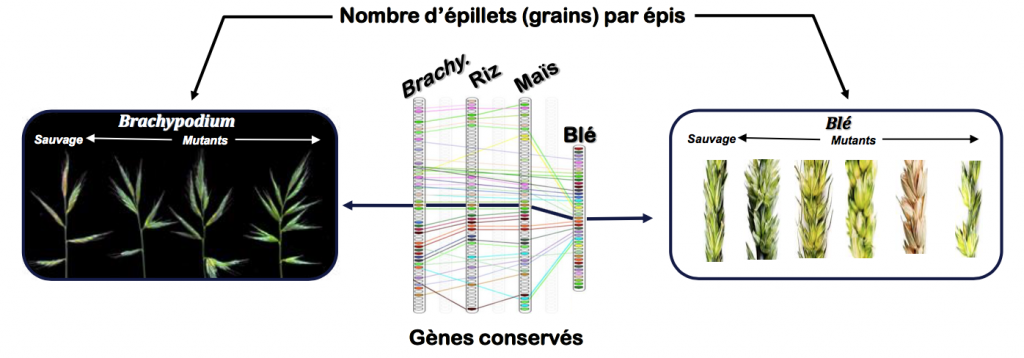
Enfin, du point de vue de la recherche appliquée, ces connaissances permettent d’identifier avec précision les régions qui portent des gènes ayant une origine commune au sein, par exemple, des génomes de riz, maïs, sorgho, brome, vigne, arabette, peuplier, papaye, luzerne, soja, pommier, concombre… Les informations connues sur la fonction biologique des gènes chez une de ces espèces permettent d’interpréter plus facilement la fonction de ces mêmes gènes chez les autres espèces apparentées. On peut donc rechercher de façon plus efficace les gènes impliqués dans les caractères d’intérêt agronomique chez une espèce et d’en étudier la fonction potentielle chez les autres espèces facilitant ainsi l’amélioration variétale : on parle de recherche translationnelle (Figure 5).
Glossaire
(1) Base : les bases, ou acides nucléiques, sont les constituants de l’ADN existantes sous 4 formes : adénine, thymine, guanine et cytosine.
(2) Gène : unité d’hérédité correspondant à un segment d’ADN et contrôlant un caractère particulier.
(3) Génome : ensemble du matériel génétique d’un individu contenu dans les cellules.
(4) Mégabase : million de bases (106).
(5) Paléogénomique : étude de la structure et de la fonction des génomes ancêtres des espèces modernes.
(6) Polyploïdie : duplication totale d’un génome par la présence dans le même noyau de plusieurs jeux de chromosomes.
(7) Synténie : conservation de groupes de gènes entre les génomes de deux espèces.
(8) Recherche translationnelle: transfert de connaissances entre espèces modèles et espèces d’intérêt agronomique ou médical.
A lire…
– Salse J (2016) Ancestors of modern plant crops. Curr Opin Plant Biol. 30:134-42.
– Salse J (2016) Deciphering the evolutionary interplay between subgenomes following polyploidy: A paleogenomics approach in grasses. Am J Bot. 103(7):1167-74.
– Pont C and Salse J (2017) Wheat paleohistory created asymmetrical genomic evolution. Curr Opin Plant Biol. 36:29-37.
Caroline Pont, Jérôme Salse
