La sélection génomique, une innovation pour l’amélioration génétique
Laurence Moreau
La sélection des espèces végétales et animales remonte à leur domestication à partir d’espèces sauvages environ - 8000 av J.-C. Suite à cette domestication, un long travail d’amélioration a été réalisé pour adapter les espèces aux différents besoins de l’homme. C’est à partir du XIXe siècle que la sélection moderne a débuté. Des méthodes de sélection ont été développées et le métier de sélectionneur a vu le jour. Ce long travail de sélection a été très efficace et a largement contribué à l’augmentation de la production agricole. Cependant, il est important de noter que jusqu’à récemment, la sélection a reposé uniquement sur l’appréciation indirecte de la valeur génétique des plantes ou des animaux au travers de leurs performances, sans connaître les gènes impliqués dans la variation observée. Ce n’est qu’à la fin des années 80 que les progrès en biologie moléculaire ont permis de mettre au point des outils révélant (ou marquant) des différences dans la séquence d’ADN d’individus à certaines positions du génome (appelées marqueurs). Très rapidement les marqueurs moléculaires ont été utilisés dans de nombreux domaines. Les applications les plus connues du grand public sont sans doute leur utilisation pour des tests de paternité ou pour l’identification par la police scientifique des auteurs de crimes à partir d’échantillons biologiques. Grâce aux outils de marquage, il a été possible d’observer dans des familles des co-transmissions entre certaines variations de séquence (ou allèle) aux marqueurs et des caractères d’intérêt comme par exemple la résistance à une maladie. Ces associations « marqueur-caractère » résultent de la proximité physique sur les chromosomes entre le marqueur considéré et un gène responsable de la variation du caractère d’intérêt. L’identification de telles associations est donc une première étape vers l’identification des zones chromosomiques porteuses de gènes d’intérêt. Outre un intérêt fondamental pour la compréhension des bases génétiques des caractères, les associations « marqueur-caractère » peuvent être utilisées directement en sélection. Chez les plantes, il est possible d’extraire l’ADN très précocement, dès le stade plantule, voire même directement au niveau de la graine. L’analyse de cet ADN permet de savoir quels sont les allèles aux marqueurs présents chez les différentes plantes et donc de prédire de façon indirecte la présence ou l’absence du/des gènes d’intérêt identifiés comme associés à ces marqueurs, bien avant que le caractère ne s’exprime au niveau de la plante. Cette sélection « assistée par marqueurs » permet donc de trier précocement les plantes porteuses des gènes d’intérêt et donc d’augmenter l’efficacité de la sélection. Cette approche s’est rapidement développée en amélioration des plantes dans les années 90 et elle est désormais utilisée en routine chez de nombreuses espèces pour améliorer des caractères « simples » qui impliquent un faible nombre de gènes. Son application reste plus difficile pour des caractères plus complexes comme le rendement où la tolérance à la sècheresse par exemple pour lesquels de nombreux gènes sont impliqués. Ces caractères sont souvent difficiles à mesurer précisément ce qui rend la recherche d’association avec les marqueurs plus difficile et moins précise.
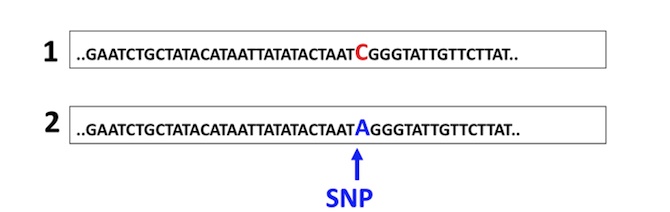
A partir des années 2000, de nombreuses initiatives internationales ont permis de séquencer le génome complet de plusieurs espèces. Chez, les plantes, l’arabette (Arabidopsis thaliana) est la première espèce à avoir été séquencée en 2000 et de nombreux génomes végétaux (riz, maïs, tomate…) ont été séquencés depuis. A l’heure actuelle, pour de nombreuses espèces d’intérêt agronomique, plusieurs génomes différents ont pu être séquencés (au moins partiellement). Ce travail de re-séquencage a permis de mettre en évidence un grand nombre de différences de séquences entre individus. Ces différences sont de deux types : des insertions-délétions de fragments de longueur variable, et des substitutions ponctuelles d’une base par une autre, parmi les quatre bases qui composent l’ADN (figure 1). Les résultats de ces études ont montré que ces différences ponctuelles (appelées SNP pour « Single Nucleotide Polymorphism ») sont extrêmement nombreuses (plusieurs millions par espèce). Même si un grand nombre de ces variations n’ont a priori pas de rôle direct dans la variabilité observée des caractères, certaines de ces variations sont causales. En parallèle à ce travail de re-séquençage, des outils se sont développés pour pouvoir caractériser (« génotyper ») simultanément un individu pour un grand nombre de SNP. Selon les techniques utilisées, on peut connaître la séquence d’un individu pour plusieurs centaines ou plusieurs centaines de milliers de SNP simultanément, grâce à des « puces » de génotypage miniaturisées (figure 2). Les progrès techniques sont tels qu’il est envisageable dans un proche avenir de pouvoir directement re-séquencer de façon plus ou moins exhaustive n’importe quel individu pour un coût modique. Ces techniques de génotypage à haute densité ouvrent des perspectives sans précédant dans de nombreux domaines puisqu’au sein des SNP se trouvent au moins en partie les variations causales des caractères.

A l’heure actuelle, de nombreux gènes ont été identifiés dans les données issues de séquençage, mais il y a très peu de cas où on a pu identifier la variation de séquence à l’origine de la variabilité d’un caractère d’intérêt. Malgré cela, sous l’hypothèse où un grand nombre de gènes sont impliqués dans les caractères, on peut penser que deux individus qui partagent un grand nombre d’allèles communs au niveau de marqueurs SNP, ont de grandes chances de partager aussi les mêmes allèles au niveau des variations causales et donc d’avoir des performances proches. C’est le principe sur lequel repose une nouvelle approche de sélection assistée par marqueurs appelée « sélection génomique » qui est en train de révolutionner le monde de la sélection. Cette approche propose d’utiliser une population dite « de référence » qui est génotypée de façon dense et qui est évaluée pour les caractères que l’on souhaite améliorer. A partir de ces données, on évalue les associations « marqueurs-caractères » non pas pour identifier de façon précise les gènes en cause mais pour établir une formule de prédiction qui permette de faire un pronostic sur la performance d’individus candidats à la sélection sur la seule base de leur génotype aux marqueurs, sans avoir besoin de les évaluer. Cette approche pose encore un grand nombre de questions scientifiques liées notamment à l’étape de calibration du modèle de prédiction (Quelle méthode statistique utiliser ? Comment choisir les individus de la population de référence ? Combien d’individus prendre ?), mais elle est dores et déjà mise en œuvre avec succès notamment pour la sélection des vaches laitières. Chez les bovins laitiers, l’effort de sélection porte surtout sur le choix de taureaux dont la semence est utilisée à grande échelle pour inséminer les vaches dans les élevages. La performance d’un taureau est évaluée au travers de la production laitière de ces filles. Pour pouvoir l’évaluer, il faut attendre que le taureau soit en âge de se reproduire et que ses filles elles-mêmes soient également en âge de se reproduire. Jusqu’en 2009, ce travail était réalisé dans des stations de « testage » dédiées et il fallait donc attendre plusieurs années avant de pouvoir évaluer précisément la valeur d’un taureau. Sachant qu’en moyenne un taureau sur dix était sélectionné, le coût de l’évaluation d’un taureau retenu était de l’ordre de 300 000 €. En 2006, le génome bovin a été séquencé, en 2008 une puce de génotypage à 50 000 SNP a été développée pour un coût d’environ 120 € par animal. La même année, la sélection génomique a été mise en place en France et en 2009, le « testage » a été abandonné. On estime que dans cette espèce la sélection génomique a permis de multiplier par deux le progrès génétique par unité de temps.
Chez les autres espèces animales et chez les plantes, de nombreux programmes de sélection génomique se mettent en place tant dans les entreprises de sélection privées qu’au niveau de la recherche publique. Plusieurs projets de recherche financés dans le cadre des « investissement d’avenir » sont développés (« Breedwheat » pour le blé, « Amaizing » pour le maïs…) et incluent un volet sur cette approche. A l’INRA, la sélection génomique fait l’objet d’un « méta-programme » dont l’objectif est d’intensifier les recherches sur ce thème au sein des différents départements de l’institut, au niveau animal, végétal mais aussi au niveau socio-économique. En effet, au-delà des aspects scientifiques, on peut s’attendre à ce que la sélection génomique conduise à des bouleversements de l’organisation de la sélection à l’instar de ce qui s’est produit chez les bovins laitiers. La création d’outils de génotypage nécessite la mise en commun de données et de moyens, ce qui n’a pu être possible chez de nombreuses espèces que grâce à la création de consortiums internationaux « privé-public ». A terme, on peut penser que cette approche permettra de faciliter le lien entre la recherche et l’application. En effet, on peut envisager de constituer des populations de référence rassemblant une large diversité génétique et de les évaluer pour des caractères particuliers (résistance à des pathogènes, test en conditions limitantes d’irrigation ou d’engrais, qualité gustative…) très difficiles à mesurer en routine dans les programmes de sélection. Après avoir calibré des équations de prédiction, il sera possible d’intégrer ces nouveaux caractères dans les programmes de sélection sans attendre d’avoir identifié précisément les gènes impliqués. De ce fait, la sélection génomique devrait permettre à l’amélioration des plantes de mieux répondre aux enjeux majeurs liés au changement climatique, de faire face aux nouvelles exigences environnementales de la société tout en répondant aux demandes des consommateurs en terme de qualité des produits.

Bonsoir, je nomme SIMO, étudiante sélectionnée, en cycle Doctorat à l’université de Yaoundé, au Cameroun. J’ai lu avec beaucoup d’intérêt votre publication, je la trouve assez édifiante. En effet, je m’intéresse à l’amélioration génétique des vaches de races locales du Cameroun. Ma motivation vient du que nos races locales ne produisent pas assez de lait contrairement aux vaches Holstein, Jersey d’Europe. Je voudrais rechercher les axes pour améliorer les performances de production de nos vaches de races locales. Cependant j’ai besoin d’une formation dans le domaine de la sélection génomique.
Une doléance, est il possible pour moi d’obtenir une formation dans ce domaine?